霍乱是一种严重的腹泻传染病,历史记录上已有七次世界大流行。时至今日,全球仍然处在第七次大流行中。霍乱由遗传学上结构单一的霍乱弧菌引起。已有研究利用基因组学分析了第七次霍乱全球大流行的扩散,定义由O1群El Tor生物型的L2谱系菌株引起,并鉴定了三个连续的亚分支,分别是Wave1、Wave2和Wave3,霍乱通过这三次流行波从南亚、东南亚多次向全球传播。但这些研究单纯使用了基因组单核苷酸多态位点(SNP)数量作为进化分枝的遗传差异界定,这导致因为水平转移的重组片段带来较多SNP位点、从而放大了遗传距离;虽然第七次大流行起自1961年,但这些分析中最早的第七次流行菌株来自1973年,也没有包括中国的霍乱流行菌株以及中亚菌株;三次流行扩散Wave也倾向于是流行病学的概念。总体上,还需要新的分析策略。
腹泻病室与爱尔兰科克大学(University College Cork)Mark Achtman教授的团队以及华大基因合作,系统选择了我国1961年到2010年期间的71株O1群霍乱流行菌株、以及来自其它国家的8株流行菌株进行了全基因组测序,与可以公开获得的另外181个霍乱弧菌基因组进行比较研究。结果显示,O1群霍乱菌株基因组的多样性主要是水平转移成分所贡献的,利用去除重组区域后的SNPs重新构建的大流行菌株的进化图提示古典型与El Tor生物型菌株之间的遗传关系并不像之前的研究提示的那样分散。结合各流行年代和地区的菌株分析,发现El Tor型霍乱弧菌在第七次流行中呈现连续累积突变的特征,之前界定的Wave相互之间在时间上出现了重叠,这也模糊了三次Wave的界限,因此,文章提出用“Clade”的概念来区分不同期的流行遗传分支,使第七次大流行菌株进化分析更为精细准确。
我国1961年开始突然出现霍乱大范围流行。对中国流行菌株的基因组分析,也显示中国流行菌株来自南亚和东南亚。60年代到90年代,多个霍乱弧菌的进化和流行分支传到中国,在中国经历进化之后,也数次传播到其它国家。因此,在第七次霍乱全球大流行中,中国既是霍乱的输入地、又是霍乱的输出地。该研究中,也从菌株基因组背景上重构了第七次大流行期间、霍乱在不同国家地区的传播关系。以上研究以“The Role of China in the Global Spread of the Current Cholera Pandemic”为题发表在2015年的PLoS Genetics杂志上。http://journals.plos.org/plosgenetics/article?id=10.1371/journal.pgen.1005072。


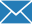
图2: 推测的霍乱在不同地区之间传播的路线
不同的色条提示不同的传播事件。色条直接接触圆圈边缘的一端代表霍乱输出地区,色条与圆圈边缘之间有空白区域的一端代表霍乱的输入地区。标记的年代为推断的传播事件发生的时间。









 2015-07-31
2015-07-31
 【打印】
【打印】

